2 무작정 해보기 #
use tempdb;
--drop table 성적
create table 성적
(
통계 int
, 결석 int
, 수학 int
);
insert 성적 values (85, 3, 65);
insert 성적 values (74, 7, 50);
insert 성적 values (76, 5, 55);
insert 성적 values (90, 1, 65);
insert 성적 values (85, 3, 55);
insert 성적 values (87, 3, 70);
insert 성적 values (94, 1, 65);
insert 성적 values (98, 2, 70);
insert 성적 values (81, 4, 55);
insert 성적 values (91, 2, 70);
insert 성적 values (76, 3, 50);
insert 성적 values (74, 4, 55);
> library("RODBC")
> conn <- odbcConnect("26")
> data <- sqlQuery(conn, "SELECT 통계, 수학, 결석 FROM tempdb.dbo.성적")
> a <- aov(통계 ~ 수학 + 결석, data)
> a
Call:
aov(formula = 통계 ~ 수학 + 결석, data = data)
Terms:
수학 결석 Residuals
Sum of Squares 541.6927 61.9657 124.5916
Deg. of Freedom 1 1 9
Residual standard error: 3.720687
Estimated effects may be unbalanced
> summary(a)
Df Sum Sq Mean Sq F value Pr(>F)
수학 1 541.69 541.69 39.1297 0.0001487 ***
결석 1 61.97 61.97 4.4762 0.0634803 .
Residuals 9 124.59 13.84
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
>
결과해석
- 가설
- 귀무가설: 요인별 차이가 없다.
- 대립가설: 요인별 차이가 있다.
- 수학의 p-value = 0.0001487로 유의수준 0.05보다 작으므로 대립가설을 뻑낼 이유를 찾지 못했다. 그러므로 수학점수와 통계점수와는 차이가 있다.
- 결석의 p-value = 0.0634803로 유의수준 0.05보다 크므로 대립가설은 뻑낼 이유를 찾았다. 그러므로 귀무가설을 지지한다. 그러므로 통계점수는 결석수에 따라서 좌지우지 된다고 할 수 있다.
3 일원 분산분석 #
c1 <- c(3.6, 4.1, 4.0)
c2 <- c(3.1, 3.2, 3.9)
c3 <- c(3.2, 3.5, 3.5)
c4 <- c(3.5, 3.8, 3.9)
data <- data.frame(c1,c2,c3,c4)
#데이터를 만들어야 한다. stack()
data <- stack(data)
summary(aov(values ~ ind, data))
결과는 다음과 같다.
Df Sum Sq Mean Sq F value Pr(>F)
ind 3 0.56250 0.18750 2.25 0.1598
Residuals 8 0.66667 0.08333
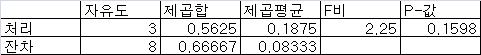
결과의 해석
- 가설
- 귀무가설: 차이가 없다.
- 대립가설: 차이가 있다.
- p-value = 0.1598 으로 대립가설이 뻑날 1종 오류의 확률이 0.05보다 크다. 그러므로 귀무가설을 지지한다.
- 즉, 3개 그룹의 평균 차이는 없다.
4 반복이 없는 다원 분산 분석 #
실험실 <- c(rep("1",4), rep("2",4), rep("3",4))
식품 <- c(rep(c("A","B","C","D"),3))
관측치 <- c(3.6,3.1,3.2,3.5,4.1,3.2,3.5,3.8,4.0,3.9,3.5,3.8)
data <- data.frame(관측치, 실험실, 식품)
data
summary(aov(관측치 ~ 식품 + 실험실), data)
결과는 다음과 같다.
> data
관측치 실험실 식품
1 3.6 1 A
2 3.1 1 B
3 3.2 1 C
4 3.5 1 D
5 4.1 2 A
6 3.2 2 B
7 3.5 2 C
8 3.8 2 D
9 4.0 3 A
10 3.9 3 B
11 3.5 3 C
12 3.8 3 D
> summary(aov(관측치 ~ 식품 + 실험실), data)
Df Sum Sq Mean Sq F value Pr(>F)
(Intercept) 1 155.520 155.520 4241.4545 8.813e-10 ***
식품 3 0.540 0.180 4.9091 0.04692 *
실험실 2 0.420 0.210 5.7273 0.04062 *
Residuals 6 0.220 0.037
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Warning messages:
1: In model.matrix.default(mt, mf, contrasts) :
variable '식품' converted to a factor
2: In model.matrix.default(mt, mf, contrasts) :
variable '실험실' converted to a factor
3: In Ops.factor(left) : ! 인자에 대해서 무의미합니다
4: In Ops.factor(left) : ! 인자에 대해서 무의미합니다
>
결과 해석
- 가설
- 귀무가설: 요인별 차이가 없다.
- 대립가설: 요인별 차이가 있다.
- 식품의 p-value = 0.04692이므로 0.05보다 작다. 즉, 대립가설을 뻑낼 건덕지를 찾지 못했다. 귀무가설 기각. 그러므로 식품별로 관측값의 평균이 차이가 있다.
- 실험실의 p-value= 0.04062이므로 0.05보다 작다. 즉, 대립가설을 뻑낼 건덕지를 찾지 못했다. 귀무가설 기각. 그러므로 실험실별로 관측값의 평균이 차이가 있다.
5 반복이 있는 다원 분산분석 #
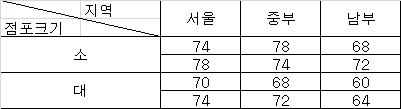
"반복이 있는"이란 뜻은 다음과 같은 뜻이다. 즉, 중복된 데이터가 있음을 뜻한다. ("select distinct 점포크기, 지역"과 "select 점포크기, 지역"은 다르다.)
지역 <-c(rep("서울",4), rep("중부",4), rep("남부",4))
점포크기 <- rep(c("소","소","대","대"), 3)
관측치 <- c(74,78,70,74,78,74,68,72,68,72,60,64)
data <- data.frame(관측치, 지역, 점포크기)
> data
관측치 지역 점포크기
1 74 서울 소
2 78 서울 소
3 70 서울 대
4 74 서울 대
5 78 중부 소
6 74 중부 소
7 68 중부 대
8 72 중부 대
9 68 남부 소
10 72 남부 소
11 60 남부 대
12 64 남부 대
반복이 있는 다원 분산분석을 실시 할 경우, 가장 먼저 교호작용이 있는지 살펴보아야 한다. 교호작용은 교호작용도(interaction plot)을 이용하면 된다. R의 경우 다음과 같이 하면 된다.
par(mfrow=c(1,2))
interaction.plot(지역,점포크기,관측치)
interaction.plot(점포크기,지역,관측치)
결과는 다음과 같다. 만약 교호작용이 발생한다면 그래프가 교차하는 부분이 생긴다.
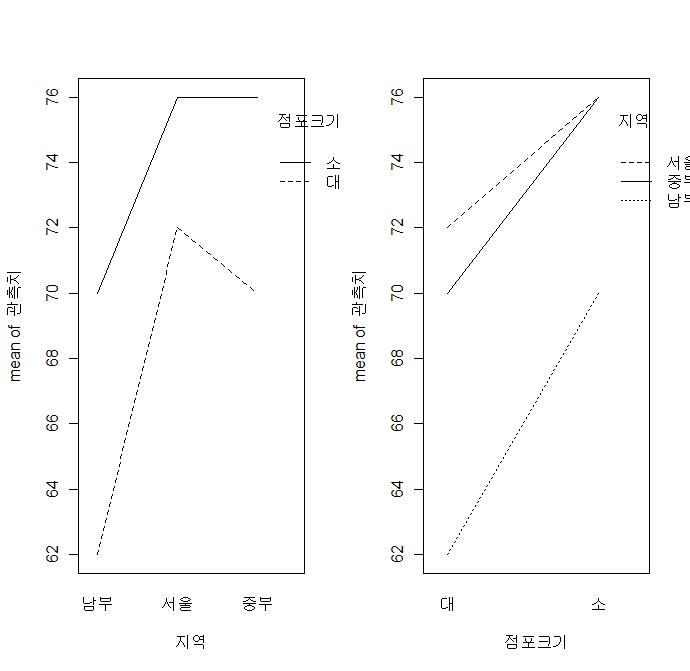
교호작용이 없는 것을 알았지만 그냥 한 번 교호작용을 하지는도 살펴보도록 하자.
summary(aov(관측치 ~ 점포크기 + 지역 + 점포크기:지역, data))
결과는 아래와 같다.
Df Sum Sq Mean Sq F value Pr(>F)
(Intercept) 1 60492 60492 7561.5 1.558e-10 ***
점포크기 1 108 108 13.5 0.01040 *
지역 2 152 76 9.5 0.01382 *
점포크기:지역 2 8 4 0.5 0.62974
Residuals 6 48 8
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Warning messages:
1: In model.matrix.default(mt, mf, contrasts) :
variable '점포크기' converted to a factor
2: In model.matrix.default(mt, mf, contrasts) :
variable '지역' converted to a factor
3: In Ops.factor(left) : ! 인자에 대해서 무의미합니다
4: In Ops.factor(left) : ! 인자에 대해서 무의미합니다
결과해석
- 가설
- 귀무가설: 요인별 차이가 없다.
- 대립가설: 요인별 차이가 있다.
- 점포크기 p-value = 0.01040 로 유의수준 0.05보다 작으므로 대립가설을 뻑낼 이유를 찾지 못했다. 대립가설을 지지하고, 귀무가설을 기각한다. 즉, 점포크기에 따라서 관측치(가격)의 차이가 있다.
- 지역 p-value = 0.01382 로 유의수준 0.05보다 작으므로 대립가설을 뻑낼 이유를 찾지 못했다. 대립가설을 지지하고, 귀무가설을 기각한다. 즉, 지역에 따라서 관측치(가격)의 차이가 있다.
- 점포크기:지역 p-value = 0.629742 로 유의수준 0.05보다 크므로 대립가설을 뻑낼 이유를 찾았다. 귀무가설을 지지한다. 즉, 점포크기와 지역의 복합효과에 대한 관측치(가격)의 차이는 없다. (교호작용 없음)
6 비모수적 방법 #
- 크루스칼-왈리스 순위합 검증(kruskcal-wallis rank sum test)
- 비모수: 소표본, 정규성 가정을 전제하기 어려울 경우
- kruskcal.test() 이용
> #세 그룹의 기관지 점막섬모의 효율성을 측정한 결과
> x <- c(2.9, 3.0, 2.5, 2.6, 3.2) #정상인
> y <- c(3.8, 2.7, 4.0, 2.4) #폐쇄성 기도질환자
> z <- c(2.8, 3.4, 3.7, 2.2, 2.0) #석면폐증 환자
> data <- data.frame(x,y,z)
이하에 에러data.frame(x, y, z) : 인수는 다른 열수를 의미합니다 5, 4
> kruskal.test(data)
Kruskal-Wallis rank sum test
data: data
Kruskal-Wallis chi-squared = 0.7714, df = 2, p-value = 0.68
>
결과해석
- 가설
- 귀무가설: 차이가 없다.
- 대립가설: 차이가 있다.
- p-value = 0.68이므로 유의수준 0.05에서 대립가설은 뻑. 그러므로 귀무가설 지지
- 즉, 3그룹의 분진제거에 대한 점막섬모 효율성은 차이가 없다.
다음과 같이 그룹을 만들어서 할 수도 있다. (g가 그룹이다)
s <- c(x,y,z)
g <- rep(1:3, c(length(x),length(y),length(z)))
data <- data.frame(s,g)
kruskal.test(s,g)
> s <- c(x,y,z)
> g <- rep(1:3, c(length(x),length(y),length(z)))
> data <- data.frame(s,g)
> kruskal.test(s,g)
Kruskal-Wallis rank sum test
data: s and g
Kruskal-Wallis chi-squared = 0.7714, df = 2, p-value = 0.68
>
7 정리중.. #
{현대실험계획법, 박성현, 민영사}의 예제 3.1
#3-1
x <- read.table(header=T, text="
factorLevel characteristicValue
A1 8.44
A1 8.36
A1 8.28
A2 8.59
A2 8.91
A2 8.6
A3 9.34
A3 9.41
A3 9.69
A4 8.92
A4 8.92
A4 8.74")
head(x)
aov.out <- aov(characteristicValue~factorLevel, data=x)
summary(aov.out)
drop1(aov.out,~.,test="F")
model.tables(aov.out, type="means", se=T)
model.tables(aov.out, type="effects", se=T)
TukeyHSD(aov.out)
plot(TukeyHSD(aov.out))
library(gplots)
plotmeans(characteristicValue~factorLevel, data=x)
lm.out <- lm(characteristicValue~factorLevel, data=x)
summary(lm.out)
summary.aov(lm.out)
#등분산 검정
bartlett.test(characteristicValue~factorLevel, data=x)
fligner.test(characteristicValue~factorLevel, data=x)
#library(HH)
#hov(characteristicValue~factorLevel, data=x)
#library(lawstat)
#levene.test(characteristicValue~factorLevel, data=x)